Rを使って鳥類などの分布図を作成する(その3:複数ページの一括出力)
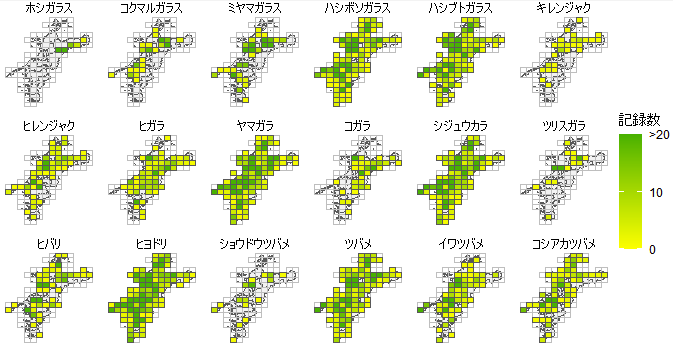
第1回目で基本地図(緯度経度・市町村別コロプレスマップ)、第2回目で地域メッシュ地図の描画方法を紹介しました。最後は地図を複数ページに分割もしくは一括出力する方法です。
野鳥情報など観察記録を扱っていると数百種類の地図を複数ページに分けてPDFなどに自動出力したい場合があると思います。数が多くなければRStudioで出力された地図を手動保存してもよいのですが、一枚一枚手動でぽちぽち保存するのは大変です。そんな面倒な処理こそ、人間ではなくパソコンに任せてしまいましょう。
ggforceパッケージを使えば、これまで紹介したコードに数行追加するだけで、ページの一括保存が簡単に実現できます。まず、複数ページに分けて地図を出力する方法です。ggforceパッケージをインストールして読み込みます。
install.packages("ggforce")
library(ggforce)そうしたら、1回目と2回目のコードで facet_wrap としていた箇所を facet_wrap_paginate に変更します。これで複数ページに分けてpage = 1 とか page = 4 などとページ数を指定して、地図を表示できるようになります。
例えば、第2回目の各種年別の分布図は、下記のコードで39地図が1ページに表示されていてちょっと窮屈でした。
facet_wrap(種名 ~ 年)これを、2行4列(1ページに8地図)で5ページに分けて、出力する図を3ページ目にしたい場合、以下のように書き換えます。
facet_wrap_paginate(種名~年, nrow = 2, ncol = 4, page = 3)全体のコードはこうなります。
kansatsu_mesh %>%
group_by(種名, メッシュ, 年) %>%
mutate(メッシュ数 = n()) %>%
ggplot() +
geom_sf(data = mapdayo, fill = "white") +
geom_sf(data = mesh10map, alpha = 0, color = "grey") +
geom_sf(aes(fill = メッシュ数, geometry = geometry)) +
scale_fill_continuous(low = "yellow", high = "#009933") +
facet_wrap_paginate(種名~年, nrow = 2, ncol = 4, page = 3) +
theme_void()RStudioの図が表示されている所の Export をクリックすれば、この図を画像やPDFとして保存できます。数ページ程度なら page の数を変えながら手動で1枚ずつ保存していってもいいのですが、一つのPDFにまとめて一括保存できれば便利ですよね。
その場合は、グラフを描画するコードの前後にこのコードを追加します。
pdf("map_zenbu.pdf", family="Japan1GothicBBB")
for (i in 1:5) {print(
#グラフを描くコード
)}
dev.off()最初の pdf( の部分では、空のPDFファイルを作成しています(map_zenbuでなく好きなファイル名でOK)。フォントも指定します。日本語環境のRなら問題ないのですが、英語のRだとフォントを指定しないと地図は出力されますが、文字化けします。
そしてその空のPDFに、次の行のfor文で、地図を1ページ目から順にプリントする処理を自動で繰り返して、という命令をしています。39地図なら1ページ8つで5ページに収まるので、1:5(1から5ページ目)としています。でも、いちいちページ数を数えるのは面倒ですよね。
ggforceでは、分割したページの合計数を数える n_pages という関数が使えます。試しに先ほどのグラフを描くコードを nanmai というデータに代入してページ数を数えてみます。
グラフを描くコードは 冒頭にnanmai <- というのを足した以外は、先ほどのものと全く同じです。
nanmai <- kansatsu_mesh %>%
group_by(種名, メッシュ, 年) %>%
mutate(メッシュ数 = n()) %>%
ggplot() +
geom_sf(data = mapdayo, fill = "white") +
geom_sf(data = mesh10map, alpha = 0, color = "grey") +
geom_sf(aes(fill = メッシュ数, geometry = geometry)) +
scale_fill_continuous(low = "yellow", high = "#009933") +
facet_wrap_paginate(種名~年, nrow = 2, ncol = 4, page = 3) +
theme_void()
n_pages(nanmai)このコードを実行すると、グラフは描画されず、コンソールに以下のように出力され、全部で5ページになることが分かります。
> n_pages(nanmai)
[1] 5この n_pages(nanmai) を使って、先ほどの1:5と同じ意味になるように、for文をこのように書き換えます。
for (i in seq_len(n_pages(nanmai))) {print(では、全部のコードを合わせてみます。facet_wrap_paginate の page = のところには for (i に合わせて、ページの数ではなくアルファベットの i を入れていることに注意してください(a でも c でも何でも動きますが慣習的に index の i が使われます)。
# ↑ 必要な前半のコードは、1回目と2回目の記事を参照してください。このコードは途中からです。
install.packages("ggforce")
library(ggforce)
nanmai <- kansatsu_mesh %>%
group_by(種名,市町,年,日) %>%
mutate(メッシュ数 = n()) %>%
ggplot() +
geom_sf(data = mapdayo, fill = "white") +
geom_sf(data = mesh10map, alpha = 0, color = "grey") +
geom_sf(aes(fill = メッシュ数, geometry = geometry)) +
scale_fill_continuous(low = "yellow", high = "#009933") +
facet_wrap_paginate(種名 ~ 年, nrow = 2, ncol = 4, page = 3) +
theme_void()
#地図を表示して出力結果を確認(3ページ目)
nanmai
#全体のページ数を確認
n_pages(nanmai)
#まずは空のPDFを作成
pdf("map_zenbudayo.pdf", family="Japan1GothicBBB")
#そのPDFに n_pages(nanmai) のページ数分プリントする
for (i in seq_len(n_pages(nanmai))) {print(
kansatsu_mesh %>%
group_by(種名, メッシュ, 年) %>%
mutate(メッシュ数 = n()) %>%
ggplot() +
geom_sf(data = mapdayo, fill = "white") +
geom_sf(data = mesh10map, alpha = 0, color = "grey") +
geom_sf(aes(fill = メッシュ数, geometry = geometry)) +
scale_fill_continuous(low = "yellow", high = "#009933") +
# page = のところは数字ではなく i とする。
facet_wrap_paginate(種名~年, nrow = 2, ncol = 4, page = i) +
theme_void()
)}
dev.off()必ず最後の dev.off() も実行するようにしてください。そうしないと、PDFのプリントが終わらず、ファイルは作成されてもエラーになってしまいます。
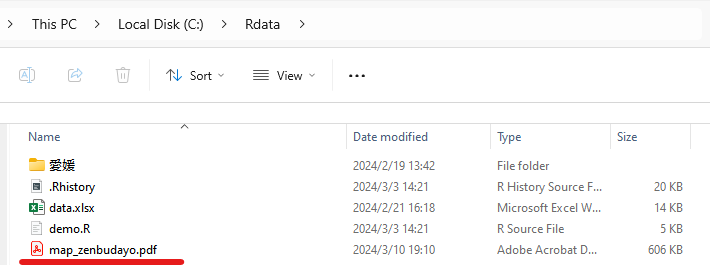
うまくいけば、このようにCドライブ直下に作成したRdataというフォルダにmap_zenbudayo.pdfというファイルが作成されていて、全ての分布図が指定した行列数(今回は2行4列)で、複数ページに分かれてこんな感じで描画されているはずです。
後は応用で、見た目や地図のカスタマイズはご自身の好みで細かく調節できますし、PDFではなく種ごとに1枚ずつ個別画像ファイルとして保存することも可能です。
Rを使ってコピペで簡単に(?)鳥類などの分布図を作成するシリーズは今回で終了です。もし蓄積はされているだけで活用されていない野鳥情報(や類似の市町村や地域メッシュ情報が含まれる生物分布情報)をお持ちでしたら、Rで可視化して、文字列だけでは見えてこなかった分布の偏りや傾向を発見してみてください。
まとめスクリプト
これまでの全てのまとめRスクリプトはGitHubに公開しています。
demo.R
おまけ:データを任意の順に並べ替える

これまでのコードで出力された図は、種名のアイウエオ順になっています。野鳥情報ならアイスランドカモメかアオアシシギが先頭に来ます。でも、分類順や記録の多い順など、任意の順番に表示したい場合があるかと思います。データに分類番号を付加してソートしたり、行番号を取得してみたり色々試したものの、なぜか数値化しても1,10,11,12…2,20,21…などと意図しない並び順になってしまい、どうすれば・・・と色々調べていたら、テキサス大のClaus O. Wilke教授の解説スライドに辿り着きました。
Getting things into the right order (最初のスライドから見る)
https://wilkelab.org/SDS375/slides/getting-things-in-order.html#62 (まとめスライド)
fct_reorder を使えばグラフの並べ替えができるようです。サンプルデータを使った場合、合計個体数が多い順(コダマ>テング>カッパ)に種名を並べる場合はこのように書きます。
kansatsu_mesh %>%
#種名だけでグループ化
group_by(種名) %>%
#種ごとに個体数の列を合計し、新たに「合計個体数」の列を作成
mutate(合計個体数 = sum(個体数)) %>%
#グループ化を一旦解除(しないと並べ替えできないです)
ungroup() %>%
#少ない順なら mutate(種名 = fct_reorder(種名, 合計個体数)) とする
mutate(種名 = fct_rev(fct_reorder(種名, 合計個体数))) %>%
ggplot() + ... 以下コード続く元データにすでに「分類番号」という列があったとして、出力される地図をその番号順に並べたい場合は以下のようになります。
motodata %>%
mutate(種名 = fct_reorder(種名, 分類番号順)) %>%
ggplot() + ... 以下コード続くこれで出力する地図の順番を自由に制御できるようになります。めでたし。



